Colistin ist ein kationisches zyklisches Peptid, das die Zellmembranen gramnegativer Bakterien zerstört. Es ist eines der wenigen verbliebenen Antibiotika der letzten Instanz, die gegen multiresistente Bakterien eingesetzt werden. Daher hat die jüngste weltweite Entdeckung mobiler Colistin-resistenter Genfamilien in einer Vielzahl multiresistenter gramnegativer Bakterien, die aus allen möglichen Umgebungen – Klinik, Veterinärmedizin, Lebensmittelindustrie und Aquakultur – isoliert wurden, Alarm geschlagen.
Der Erfolg von mcr-1 als transformierender Resistenzfaktor bleibt jedoch rätselhaft, da seine Expression die Wachstumseigenschaften von Bakterien selektiv benachteiligt und nur mäßige Resistenzniveaus gegenüber Colistin verleiht.
Nun hat ein internationales Team unter der Leitung von Wissenschaftlern des Deutschen Zentrums für Infektionsforschung (DZIF) herausgefunden, warum mcr-1 – trotz seiner Mängel – für Bakterien nützlich ist. Ihre Studie legt nahe, dass der Erwerb von mcr-1 einen spezifischen physiologischen Zustand bei Bakterien induziert, der die Widerstandsfähigkeit gegenüber häufigen Umweltstressbedingungen wie Veränderungen des Säuregehalts und antimikrobiellen Peptiden fördert.
„Wir haben herausgefunden, dass Bakterien, die mcr-1 beherbergen, regulatorische Komponenten der Stressreaktion der Bakterienhülle auslösen, ein System, das Schwankungen in der Nährstoffverfügbarkeit und Umweltveränderungen erkennt. Dies wiederum erhöht die mcr-1-Produktion dramatisch und verbessert das Überleben von Bakterien in niedrigen.“ pH-Umgebungen“, sagt er. Erstautorin der Arbeit ist Dr. Renate Frantz von der Abteilung für Medizinische Mikrobiologie der Justus-Liebig-Universität Gießen.
Die Ergebnisse deuten darauf hin, dass die Integration der MCR-1-abhängigen Resistenzaktivität in die Stressreaktion der Hülle die Resistenz von Stämmen in stressigen Umgebungen für Bakterienzellen unterstützen würde, beispielsweise während der Passage durch den Magen-Darm-Trakt oder bei Exposition gegenüber Gallensäuren.
„Unsere Analysen zeigten außerdem, dass die Produktion des MCR-1-Proteins in Bakterien, die unter leicht sauren Bedingungen wachsen, zu einer erhöhten Modifikation von Lipid A führt, der Primärstruktur von Lipopolysacchariden – einem wichtigen Bestandteil der Bakterienmembran, die für die Colistin-Resistenz erforderlich ist“, sagt Dr . Nicolas Gisch, gemeinsamer Erstautor vom Forschungszentrum Boerstel, Leibniz-Lungenzentrum.
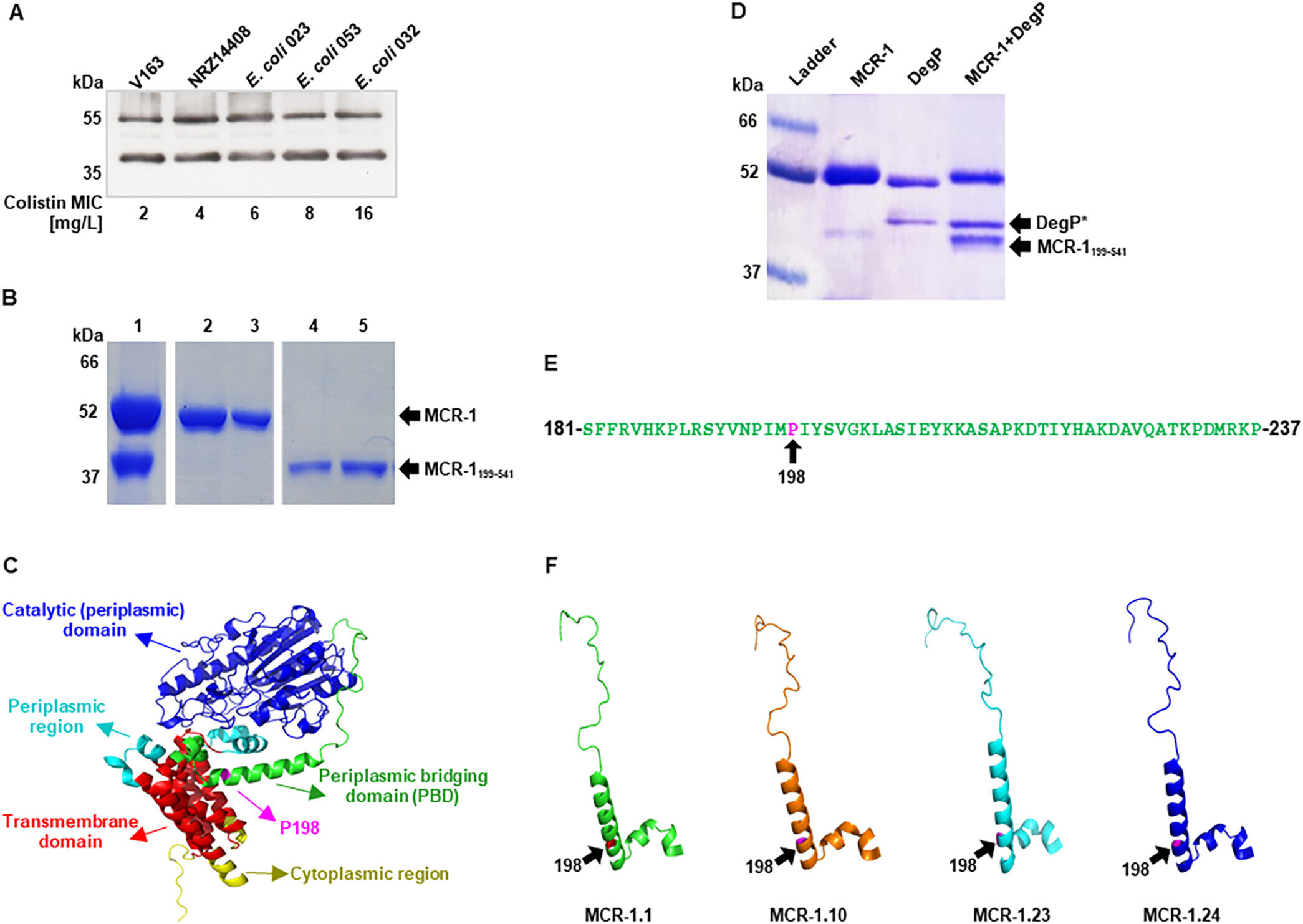
Basierend auf der Erkenntnis, dass die Aktivität des MCR-1-Enzyms unter sauren Bedingungen deutlich erhöht ist, entwickelte das Forschungsteam einen einfachen und leicht reproduzierbaren Test zur Bestimmung der MCR-1-abhängigen Colistin-Resistenz in Bakterienisolaten.
Die Produktion des MCR-1-Proteins stimuliert auch die Expression von DegP, einer Protease im Periplasma von Bakterien (dem Raum zwischen der inneren und äußeren Membran bei gramnegativen Bakterien), die MCR-1 an einer bestimmten Stelle innerhalb einer hochkonservierten Region spaltet des Proteins. „Die Modifikation der MCR-1-Spaltstelle hat tiefgreifende Auswirkungen sowohl auf die Resistenzaktivität als auch auf die Auslösung der Hüllstressreaktion“, fügt Co-Hauptautor Dr. Konrad Guzdzinski, ehemaliger DZIF-Forscher und jetzt leitender Wissenschaftler bei Mondelēz International, hinzu.
Diese Erkenntnisse über die biomolekularen Grundlagen der MCR-1-abhängigen Resistenz ermöglichten es dem Team, eine allgemeine Strategie zu entwickeln, die durch gezielte Aktivierung von Proteasen mcr-1-tragende Plasmide aus bakteriellen Wirten eliminiert.
Kommentare Professor Trinad Chakraborty, ehemaliger Direktor des Instituts für Medizinische Mikrobiologie der Justus-Liebig-Universität, der die Studie leitete. „Unsere Daten ermöglichten es uns auch, einen neuen Ansatz zur Eliminierung der übertragbaren Colistin-Resistenz bei gramnegativen Bakterien zu entwickeln, um ihrer Ausbreitung und Verbreitung in der Umwelt entgegenzuwirken.“
Veröffentlichung der Forschung in der Zeitschrift Spektrum-Mikrobiologie.
Mehr Informationen:
Renate Frantz et al., Ein einzelner Rest innerhalb des MCR-1-Proteins verleiht prädiktive Flexibilität, Spektrum-Mikrobiologie (2023). doi: 10.1128/spect.03592-22
Bereitgestellt vom Deutschen Zentrum für Infektionsforschung

„Unapologetic Analyst. Wütend bescheidener Kaffee-Evangelist. Gamer. Kann nicht mit Boxhandschuhen tippen. Student. Unternehmer.“









More Stories
Die deutsche Luft- und Raumfahrtindustrie steht vor dem Ende des Typhoon-Programms
Airbus erhält 2,1 Milliarden Euro für den Aufbau eines deutschen militärischen Kommunikationssystems
RFA, ATMOS und Yuri kündigten den Mikrogravitationsdienst „Eva“ an